大規模観測データ解析システム 利用手引き
英語(English)
作成: 2019 Apr. 9
最終更新: 2026 Jan. 22
目次:
0: 本手引きの目的:
1: システム情報:
2: 利用申請:
2-1: 利用申請(HSC共同利用観測者向け):
2-2: 利用申請(一般向け):
3: 本システムの利用方法:
4: ログインノードへのログイン方法と利用上の注意:
5: ホーム領域:
6: 大容量作業領域(/gpfs):
7: キュー構成:
8: ジョブ投入方法:
8-1: hscPipe使用時:
8-2: hscPipe以外:
9: 導入ソフト一覧:
10: hscPipeの利用準備:
11: 多波長データ解析システムとのファイルコピー:
12: 複数のユーザ間でのデータ共有:
13: ヘルプデスク:
0: 本手引きの目的:
本手引きは、大規模観測データ解析システム(以下大規模解析システム)の
基本的な利用方法と、HSCのデータ解析パイプライン hscPipe を
本システムでジョブ管理システムの元実行するための方法を紹介するものです。
多波長データ解析システムの詳細については、多波長データ解析システムを、
HSCや hscPipe についてはHyper Suprime-Camをご参照ください。
なお、ユーザ権限でhscPipeをインストールした場合には、「9:導入ソフト一覧の補足(の2番目)」を
参照した上で、そこに提示されているコマンド群を「必ず」実行してください。
1: システム情報:
大規模観測データ解析システム:
ログインノード: 1台 lsc.ana.nao.ac.jp
OS: RHEL7
計算ノード: 40台 総CPUコア数: 2,296 2,240,
39台 総メモリ量: 24.5TB 23.5TB,
OS: RHEL7/CentOS7 → CentOS7 (2024/04/04-)
ホーム領域: 36TB 1ユーザ当たり200GBの使用制限あり
大容量作業領域: 5PB /gpfs ファイルシステム: IBM Storage Scale, 1ユーザ当たり30TBの使用制限あり
ジョブ管理: PBS Professional Opensource版
スペック詳細:
- ログインノード:
CPU: Intel(R) Xeon(R) Silver 4114 2.2GHz 10core x2
Memory: 256GB
OS: Red Hat Enterprise Linux 7.9
- 計算ノード:
- an[01-02*]
CPU: Intel(R) Xeon(R) Gold 6132 2.6GHz 14core x4
Memory: 1TB (PBS利用可:950GB)
OS: Red Hat Enterprise Linux 7.6 → CentOS Linux 7.6 (2023/09/21-) → 7.7 (2025/03/04-) → 7.9 (2025/09/01-)
(* an02は故障のため2025/04/07より運用停止中)
- an[03-05]
CPU: Intel(R) Xeon(R) Gold 6132 2.6GHz 14core x4
Memory: 1TB (PBS利用可:950GB)
OS: Red Hat Enterprise Linux 7.6 → CentOS Linux 7.6 (an0[3,4]:2023/11/15-, an05:2024/04/04-) → 7.7 (2025/03/04-) → 7.9 (2025/09/01-)
- an[06-07]
CPU: AMD EPYC 7601 2.2GHz 32core x2
Memory: 512GB (PBS利用可:455GB)
OS: CentOS Linux 7.6 → 7.7 (2025/03/04-) → 7.9 (2025/09/01-)
- an[08-29]
CPU: AMD EPYC 7742 2.25GHz 64core
Memory: 512GB (PBS利用可:455GB)
OS: CentOS Linux 7.7 → 7.9 (2025/09/01-)
- an[30-36]
CPU: AMD EPYC 7742 2.25GHz 64core
Memory: 1024GB (PBS利用可:940GB)
OS: CentOS Linux 7.7 → 7.9 (2025/09/01-)
- an[91-94]
CPU: Intel(R) Xeon(R) W-2145 3.70GHz 8core
Memory:
an9[1-2,4]: 128GB + Swap:2,048GB(SSD) (PBS利用可:1,990GB)
an93: 128GB + Swap:2,000GB(SSD) (PBS利用可:1,940GB)
OS: CentOS Linux 7.7 → 7.9 (2025/09/01-)
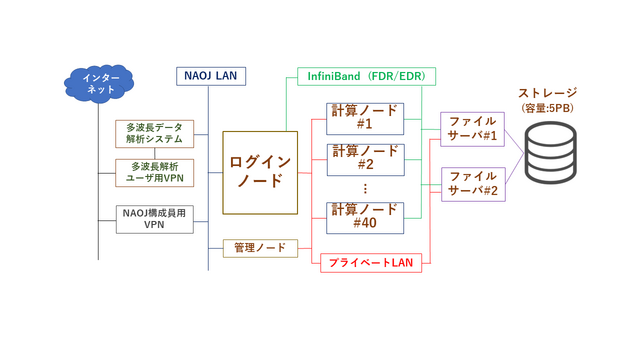
システム構成図:
 2: 利用申請:
本システムの利用申請は、利用希望者が当該セメスターのHSC共同利用観測者
(PI+CoI、インテンシブを含む)かどうかで申請方法が異なります。
御自身が該当する方を御参照ください。
2-1: 利用申請(HSC共同利用観測者向け):
対象課題のPIには、セメスター開始前に、ハワイ観測所からLSCについての
ご案内をお送りします。
LSCの利用手順は、観測所からの案内をご参照ください。
また、HSC共同利用観測者向けのLSC利用に関するお問い合わせは、
ハワイ観測所のLSC担当(daikibo [at-mark] naoj.org)へお送りください。
2-2: 利用申請(一般向け):
随時利用申請を受け付けております。
利用申請ページ(一般向け)より申請をおこなってください。
3: 本システムの利用方法:
大規模解析システムは、計算資源をジョブ管理システムで管理します。
ユーザはログインノードにログインし、ジョブを投入することで
計算ノードで解析処理を実行可能です。
計算ノードへのログインや、システムに定められた目的以外の利用
およびジョブ管理システムを介さない利用は認めていません。
そのような利用を発見した場合には、予告なく強制ログアウト、
プロセスの強制停止を行いますのでご了承ください。
4: ログインノードへのログイン方法と利用上の注意:
天文台内からであれば、ログインノードへの ssh でログイン可能です。
ログインノードのFQDN: lsc.ana.nao.ac.jp
天文台外からであれば、NAOJ構成員用のVPNまたは多波長データ解析システムの
VPNで台内に接続後、 ログインノードへの ssh でログイン可能です。
アカウントは、多波長データ解析システムのアカウントを使用します。
(パスワードも同一)
大規模解析システムは、多波長データ解析システムのアカウント情報を共有しています。
ただし、これら登録情報の変更は大規模解析システム上で行うことが出来ません。
変更は、多波長データ解析システムで行うようにしてください。
2025年12月より、ログインノードの計算資源に以下の使用制限を導入しています。
- CPU使用率: ユーザ当たり200% (2コア フル稼働に相当)
- メモリ量: ユーザ全体で125G
ログインノードではジョブ投入やその準備など軽い処理のみ行なうこととし、多くの
計算資源を必要とする処理は計算ノードで行なうようにしてください。
5: ホーム領域:
ホーム領域には、200GBの使用制限(クォータ)がかかっています。
解析用のデータは、大容量作業領域(/gpfs)をご利用ください。
ホーム領域の使用量とクォータ情報は、ログインノードで
quota (-s)
の実行で確認可能です。(-sオプション: 量の単位を自動調整)
(領域名が/homeではなく「nas01:/mnt/nas/LUN_NAS」と表示される)
6: 大容量作業領域(/gpfs)の利用方法:
大容量作業領域(/gpfs)に、ご自身のアカウント名のディレクトリを
作成し、ご利用ください。
本領域は全ログイン・計算ノードから読み書き可能な領域で、30TB
の使用制限(クォータ)がかかっています(2025/6/2より)。
(定期)データ削除は「現状」では導入していませんが
今後の利用状況によっては導入の可能性があります。
現在のgpfs領域の使用量は
/gpfs/usage_info/[アカウント名].txt
で確認することが出来ます。この情報は毎時0分に更新されます。
7: キュー構成:
2: 利用申請:
本システムの利用申請は、利用希望者が当該セメスターのHSC共同利用観測者
(PI+CoI、インテンシブを含む)かどうかで申請方法が異なります。
御自身が該当する方を御参照ください。
2-1: 利用申請(HSC共同利用観測者向け):
対象課題のPIには、セメスター開始前に、ハワイ観測所からLSCについての
ご案内をお送りします。
LSCの利用手順は、観測所からの案内をご参照ください。
また、HSC共同利用観測者向けのLSC利用に関するお問い合わせは、
ハワイ観測所のLSC担当(daikibo [at-mark] naoj.org)へお送りください。
2-2: 利用申請(一般向け):
随時利用申請を受け付けております。
利用申請ページ(一般向け)より申請をおこなってください。
3: 本システムの利用方法:
大規模解析システムは、計算資源をジョブ管理システムで管理します。
ユーザはログインノードにログインし、ジョブを投入することで
計算ノードで解析処理を実行可能です。
計算ノードへのログインや、システムに定められた目的以外の利用
およびジョブ管理システムを介さない利用は認めていません。
そのような利用を発見した場合には、予告なく強制ログアウト、
プロセスの強制停止を行いますのでご了承ください。
4: ログインノードへのログイン方法と利用上の注意:
天文台内からであれば、ログインノードへの ssh でログイン可能です。
ログインノードのFQDN: lsc.ana.nao.ac.jp
天文台外からであれば、NAOJ構成員用のVPNまたは多波長データ解析システムの
VPNで台内に接続後、 ログインノードへの ssh でログイン可能です。
アカウントは、多波長データ解析システムのアカウントを使用します。
(パスワードも同一)
大規模解析システムは、多波長データ解析システムのアカウント情報を共有しています。
ただし、これら登録情報の変更は大規模解析システム上で行うことが出来ません。
変更は、多波長データ解析システムで行うようにしてください。
2025年12月より、ログインノードの計算資源に以下の使用制限を導入しています。
- CPU使用率: ユーザ当たり200% (2コア フル稼働に相当)
- メモリ量: ユーザ全体で125G
ログインノードではジョブ投入やその準備など軽い処理のみ行なうこととし、多くの
計算資源を必要とする処理は計算ノードで行なうようにしてください。
5: ホーム領域:
ホーム領域には、200GBの使用制限(クォータ)がかかっています。
解析用のデータは、大容量作業領域(/gpfs)をご利用ください。
ホーム領域の使用量とクォータ情報は、ログインノードで
quota (-s)
の実行で確認可能です。(-sオプション: 量の単位を自動調整)
(領域名が/homeではなく「nas01:/mnt/nas/LUN_NAS」と表示される)
6: 大容量作業領域(/gpfs)の利用方法:
大容量作業領域(/gpfs)に、ご自身のアカウント名のディレクトリを
作成し、ご利用ください。
本領域は全ログイン・計算ノードから読み書き可能な領域で、30TB
の使用制限(クォータ)がかかっています(2025/6/2より)。
(定期)データ削除は「現状」では導入していませんが
今後の利用状況によっては導入の可能性があります。
現在のgpfs領域の使用量は
/gpfs/usage_info/[アカウント名].txt
で確認することが出来ます。この情報は毎時0分に更新されます。
7: キュー構成:
| Queue name |
Priority |
Number of available
CPU cores |
Amount of available
memory(unit:GB) |
Number of executable jobs |
Maximum Wall time |
Execution node |
| Max. |
default |
Max. |
default |
Hard |
Soft |
| qssp |
very high |
1,536 |
64 |
13,950 |
11,250 |
--- |
--- |
1,000 days |
an[13-36] |
| qh |
high |
640 |
56 |
4,500 |
450 |
--- |
--- |
1,000 days |
an[01-11,21-36] |
| qm |
medium |
112 |
56 |
1,800 |
450 |
2 |
1 |
15 days |
an[01-11] |
| ql |
low |
32 |
28 |
450 |
225 |
2 |
1 |
7 days |
an[01-11] |
| qt |
very high |
4 |
4 |
64 |
64 |
1 |
1 |
10 minutes |
an[01-11] |
| qhm |
medium |
32 |
8 |
7,910 |
1,940 |
--- |
1 |
15 days |
an[91-94] |
補足:
- 利用可能なキューは、ユーザ階層によって異なります。
* qssp は HSC-SSP (HSCを用いたハワイ観測所戦略枠観測プログラム)
解析専用キューです。
* HSC共同利用観測者は、「現状では」 qm, qt, qhm を利用可能です。
* 一般ユーザは、「現状では」 ql, qt, qhm を利用可能です。
- qt キューはジョブスクリプトの動作確認など、テスト専用キューです。
- qhm キューは他の計算ノードでは実行できない、1プロセスで1TB超の
メモリを必要とするような解析を行うためのキューです。
qhm キューを使用するジョブは、2TBのswap設定ノード(an[91-94])で
のみ実行されます。
- キューの割当計算資源・実行可能ジョブ数は利用状況に応じて変更の
可能性があります。
8: ジョブ投入方法:
ジョブ投入方法をhscPipe使用の有無で分けて紹介します。
PBSコマンドやPBS指示文、PBS環境変数を含む PBS Professional の詳細については、
旧・多波長データ解析システムの「ユーザーズガイド 4.2.4 節」 をご参照ください。
8-1: hscPipe使用時:
hscPipeの各処理を計算ノードで実行するにはジョブスクリプトを
用意し、qsubコマンドでジョブとして投入する必要があります。
hscPipeの各処理で、ジョブスクリプトを作成する手順は、オプションに
「--batch-type」を持つコマンドと持たないコマンドで異なります。
A: optionに「--batch-type」を持つコマンド:
1. 各処理の実行時に
「--batch-type=pbs --time 100000 --nodes 2 --procs 112 --dry-run」
を追加して実行する。
(時間やノード数、CPUコア数は適当で良い。3で変更可能なため)
2. 出力される一時ファイルを作業領域に別名コピー。
3. 2でコピーしたファイルを開き、以下の編集を行う
3-1: PBS指示文の追加:
冒頭のPBS指示文を適切な指示文に編集し、必要なPBS指示文があれば
追加する。
例1: (キュー:qm、計算資源として「計112コア・メモリ1792GB」を使用する場合)
#PBS -q qm (キュー:qmを使用する)
#PBS -m a (ジョブが中止された際にメール送信)
#PBS -l select=112:ncpus=1:mpiprocs=1:mem=16g
(使用資源の制限:1コア当たり16GBのメモリを計112コア分使用)
#PBS -M 送信先メールアドレス(※8-1-1)
#PBS -N qm_CB (ジョブに名前「qm_CB」をつける)
#PBS -l walltime=24:00:00 (最大実行時間の指定:24時間)
#PBS -o log_qsub_CB.out (標準出力のファイル出力先:log_qsub_CB.out)
#PBS -e log_qsub_CB.err (標準エラー出力のファイル出力先:log_qsub_CB.err)
補足: 必要とする計算資源に応じた「#PBS -l」の記述例:
- 計算資源として「計112コア・メモリ896GB (コア当たり8GB)」を使用する場合、
#PBS -l select=112:ncpus=1:mpiprocs=1:mem=8g
- 計算資源として「計28コア・メモリ448GB (コア当たり16GB)」を使用する場合、
#PBS -l select=28:ncpus=1:mpiprocs=1:mem=16g
例2: (キュー:ql、計算資源として「計28コア・メモリ448GB」を使用する場合)
#PBS -q ql (キュー:qlを使用する)
#PBS -m a (ジョブが中止された際にメール送信)
#PBS -l select=28:ncpus=1:mpiprocs=1:mem=16g
(使用資源の制限:1コア当たり16GBのメモリを計28コア分使用)
#PBS -M 送信先メールアドレス(※8-1-1)
#PBS -N ql_CB (ジョブに名前「ql_CB」をつける)
#PBS -l walltime=24:00:00 (最大実行時間の指定:24時間)
#PBS -o log_qsub_CB.out (標準出力のファイル出力先:log_qsub_CB.out)
#PBS -e log_qsub_CB.err (標準エラー出力のファイル出力先:log_qsub_CB.err)
例3: (キュー:ql、計算資源として「計32コア・メモリ448GB」を使用する場合)
例2の「#PBS -l select...」行を以下に置き換える。それ以外の行は例2と同じ
#PBS -l select=32:ncpus=1:mpiprocs=1:mem=14g
(使用資源の制限:1コア当たり14GBのメモリを計32コア分使用)
※8-1-1: 「送信先メールアドレス」にgmailのアドレスを使用するのは極力避ける。
gmail宛メールの送信には現在強い制限をかけているので、大量のメール送信は
ジョブメールの送信遅延を引き起こす。
gmail宛以外のメールに対してもゆるやかな送信制限をかけてるので、短期間に
大量のメールが送信されないよう、投入するジョブ数に応じて適切な通知設定を行う。
3-2: mpiexec行の編集:
mpiexec行の、python以降を別ファイルに記述し、ファイルに実行権限を付与。
mpiexec行の引数にファイルをフルパスで指定する。
一例: constructBias.pyの場合:
mpiexec python -c 'import os; os.umask(0o002); ...
のpython以降をファイル:exec_CB.shに記述、実行権限を付与:
chmod u+x exec_CB.sh
cat exec_CB.sh
-> python -c 'import os; os.umask(0o002); ...
mpiexecの引数としてフルパスでファイルを指定したものに置換する
mpiexec /パス/exec_CB.sh
参考: 上記を実行するコマンド: (hscPipe7での動作確認済み)
$ dirb=$(pwd) # ファイルの在り処(任意:ここではカレントディレクトリ)
$ fnqs=qsub_CB.sh # 編集するジョブスクリプト名(任意)
$ pyfn=exec_CB.sh # python以降を書き込むファイル名(任意)
$ grep ^mpiexec ${fnqs} | sed -e "s/mpiexec\ \+python/python/" > ${pyfn}
$ chmod u+x ${pyfn}
$ sed -i.bak "/mpiexec/s|python .\+$|${dirb}/${pyfn}|" ${fnqs}
← 編集前のファイルが、「$fnqs}.bak」として残る
(残さずに上書きする場合は 「-i.bak」 を 「-i 」とする)
4. 3で作成したジョブスクリプトをqsubコマンドの引数として
与え、実行する。
qsub -V ジョブスクリプト名
B: optionに「--batch-type」を持たないコマンド:
(例: mosaic.py や jointcal.py)
1. ジョブスクリプトを1から作成する。エディタで新規ファイルを開き、
1行目にshellのシェバン(例:「#!/usr/bin/env bash」)、2行目以降に
PBS指示文を記述する(8-1のA-3を参照)。
補足: 必要とする計算資源に応じた「#PBS -l」の記述例:
(8-1のA-3-1とは異なり、1つのノード上で実行するための記述)
- 計算資源として「1ノード、計64コア・メモリ450GB」を使用する場合、
#PBS -l select=1:ncpus=64:mpiprocs=64:mem=450g
- 計算資源として「1ノード、計28コア・メモリ225GB」を使用する場合、
#PBS -l select=1:ncpus=28:mpiprocs=28:mem=225g
2. PBS指示文のあとは hscPipe の利用準備コマンド群(「10: hscPipeの利用準備」を参照)を記述。
その後に、ディレクトリの移動コマンドと実行するコマンドを記述する。
3. 2で作成したジョブスクリプトをqsubコマンドの引数として
与え、実行する。
qsub -V ジョブスクリプト名
8-2: hscPipe以外:
hscPipe以外の場合でも、ジョブスクリプトを用意し、qsubコマンドで
ジョブとして投入する必要があります。
1. ジョブスクリプトを1から作成する。エディタで新規ファイルを開き、
1行目にshellのシェバン(例:「#!/usr/bin/env bash」)、2行目以降に
PBS指示文を記述する(8-1のA-3を参照)。
補足: 必要とする計算資源に応じた「#PBS -l」の記述例:
- 計算資源として「計28コア・メモリ448GB (コア当たり16GB)」を使用する場合、
(複数の計算ノードが実行ノードになる可能性あり)
#PBS -l select=28:ncpus=1:mpiprocs=1:mem=16g
- 計算資源として「1ノード、計28コア・メモリ225GB」を使用する場合、
(実行ノードは1台の計算ノードのみ)
#PBS -l select=1:ncpus=28:mpiprocs=28:mem=225g
2. PBS指示文の後に、ディレクトリの移動コマンドと実行するコマンドを記述する。
3. 2で作成したジョブスクリプトをqsubコマンドの引数として
与え、実行する。
qsub -V ジョブスクリプト名
9: 導入ソフト一覧:
ソフト名 バージョン パス
- hscPipe 4.0.5 /opt/hscpipe/4.0.5/(bashrc) ※1
5.4 /opt/hscpipe/5.4/(bashrc)
6.7 /opt/hscpipe/6.7/(bashrc)
7.9.1 /opt/hscpipe/7.9.1/(bashrc)
8.4 /opt/hscpipe/8.4_anaconda/(loadLSST.bash)
8.5.3 /opt/hscpipe/8.5.3_anaconda/(loadLSST.bash)
- ds9 8.0.1 /usr/local/bin/ds9
- fv 5.5 /usr/local/fv/fv
- WCSTools 3.9.5 /usr/local/WCSTools/bin
- gnuplot 5.2.7 /usr/local/bin/gnuplot
- mpich 3.2.1 /usr/local/mpich/3.2.1
- PBS Pro. 19.1.2 /opt/pbs
※1 hscPipe 4.0.5のアストロメトリ用カタログファイルは未導入です。
補足:
- ユーザ権限でインストール可能なソフトウェアについては、本システムの
共有領域(/home や /gpfs)へインストールすることも可能です。
システム領域へのインストールが必要なライブラリなどがある場合には
ヘルプデスクへお問い合わせください。
- hscPipeをユーザ権限でインストールした場合には、インストール後に
以下のコマンド群を実行してください。
(これを実行しないと、ジョブ管理ソフトはユーザ権限でインストール
したhscPipeを使用するジョブを適切に制御出来ません)
bash (デフォルトシェルがbash以外の場合のみ実行)
hscPipeの実行準備
(例えばビルド版であれば、以下の実行:
source /path/to/hscPipe/loadLSST.bash
setup hscPipe バージョン番号)
com=$(which mpiexec); comd=${com%/*}; echo ${com}; echo ${comd}
cp -a ${comd} ${comd}.org
mv -v ${com} ${com}.actual
ln -s /opt/pbs/bin/pbsrun.mpich ${com}
ls -l ${com} ${com}.actual
-> mpiexecが/opt/pbs/bin/pbsrun.mpichへのリンクに、
また、mpiexec.actualがmpiexec.hydraへのリンクになっていれば良い。
(ログインノードには 「/opt/pbs/bin/pbsrun.mpich」が存在しないため
リンク切れと表示されるが、計算ノードでは存在しているので問題なし。)
10: hscPipeの利用準備:
10.1: hscPipe 4.0.5, 5.4, 6.7, 7.9.1の場合※2
unset LD_LIBRARY_PATH
source /opt/hscpipe/{4.0.5,5.4,6.7,7.9.1}/bashrc
setup-hscpipe
※2 hscPipe 7.9.1でmosaic.py を使用する場合には、使用前に
$ export LD_LIBRARY_PATH="${LD_LIBRARY_PATH}:/opt/hscpipe/7.9.1/lib64"
の実行が必要です(上記はbashの場合)。
10.2: hscPipe 8.4, 8.5.3の場合
unset LD_LIBRARY_PATH
source /opt/hscpipe/{8.4,8.5.3}_anaconda/loadLSST.bash
setup hscPipe {8.4,8.5.3}
11: 多波長データ解析システムとのファイルコピー:
多波長データ解析システムとのファイルコピーは、scpやrsyncコマンドの実行で
実現可能です。
直接マウントした「lsc:/lwk」領域も利用可能ですが、ファイルやディレクトリの
修正日時(mtime)が状態変更日時(ctime)に変わってしまう問題が確認されているため(※3)、
正しい修正日時を保持したコピーを希望する場合には「lsc:/lwk」の利用を推奨しません。
※3 タイムスタンプを保存するオプションを使用した場合でも、この問題が起こります。
発生確率は一定ではありませんが、これまでの調査では 数%程度 でした。
例: mana01の/home/username/filename -> /gpfs/username/以下へのコピー
scp -p mana01:/home/username/filename /gpfs/username/
12: 複数のユーザ間でのデータ共有:
本システムでも多波長データ解析システムのグループIDを利用可能です。
この機能を利用すると、同じ研究グループのメンバーなど、複数のユーザ間で
データファイルを共有することが可能です。
グループIDの申請は、多波長データ解析システムのグループID利用申請ページ
より行ってください。
本システムでのグループIDの利用方法は、多波長データ解析システムでの方法と
同じですので、多波長データ解析システムのグループID 利用手引きをご参照
ください。
13: ヘルプデスク:
本システムのhscPipe及びHSCデータ解析に関するお問い合わせは、
helpdesk [at-mark] hsc-software.mtk.nao.ac.jp
へご連絡ください。
上記以外に関するお問い合わせは、
lsc-consult [at-mark] ana.nao.ac.jp
へご連絡ください。
 2: 利用申請:
本システムの利用申請は、利用希望者が当該セメスターのHSC共同利用観測者
(PI+CoI、インテンシブを含む)かどうかで申請方法が異なります。
御自身が該当する方を御参照ください。
2-1: 利用申請(HSC共同利用観測者向け):
対象課題のPIには、セメスター開始前に、ハワイ観測所からLSCについての
ご案内をお送りします。
LSCの利用手順は、観測所からの案内をご参照ください。
また、HSC共同利用観測者向けのLSC利用に関するお問い合わせは、
ハワイ観測所のLSC担当(daikibo [at-mark] naoj.org)へお送りください。
2-2: 利用申請(一般向け):
随時利用申請を受け付けております。
利用申請ページ(一般向け)より申請をおこなってください。
3: 本システムの利用方法:
大規模解析システムは、計算資源をジョブ管理システムで管理します。
ユーザはログインノードにログインし、ジョブを投入することで
計算ノードで解析処理を実行可能です。
計算ノードへのログインや、システムに定められた目的以外の利用
およびジョブ管理システムを介さない利用は認めていません。
そのような利用を発見した場合には、予告なく強制ログアウト、
プロセスの強制停止を行いますのでご了承ください。
4: ログインノードへのログイン方法と利用上の注意:
天文台内からであれば、ログインノードへの ssh でログイン可能です。
ログインノードのFQDN: lsc.ana.nao.ac.jp
天文台外からであれば、NAOJ構成員用のVPNまたは多波長データ解析システムの
VPNで台内に接続後、 ログインノードへの ssh でログイン可能です。
アカウントは、多波長データ解析システムのアカウントを使用します。
(パスワードも同一)
大規模解析システムは、多波長データ解析システムのアカウント情報を共有しています。
ただし、これら登録情報の変更は大規模解析システム上で行うことが出来ません。
変更は、多波長データ解析システムで行うようにしてください。
2025年12月より、ログインノードの計算資源に以下の使用制限を導入しています。
- CPU使用率: ユーザ当たり200% (2コア フル稼働に相当)
- メモリ量: ユーザ全体で125G
ログインノードではジョブ投入やその準備など軽い処理のみ行なうこととし、多くの
計算資源を必要とする処理は計算ノードで行なうようにしてください。
5: ホーム領域:
ホーム領域には、200GBの使用制限(クォータ)がかかっています。
解析用のデータは、大容量作業領域(/gpfs)をご利用ください。
ホーム領域の使用量とクォータ情報は、ログインノードで
quota (-s)
の実行で確認可能です。(-sオプション: 量の単位を自動調整)
(領域名が/homeではなく「nas01:/mnt/nas/LUN_NAS」と表示される)
6: 大容量作業領域(/gpfs)の利用方法:
大容量作業領域(/gpfs)に、ご自身のアカウント名のディレクトリを
作成し、ご利用ください。
本領域は全ログイン・計算ノードから読み書き可能な領域で、30TB
の使用制限(クォータ)がかかっています(2025/6/2より)。
(定期)データ削除は「現状」では導入していませんが
今後の利用状況によっては導入の可能性があります。
現在のgpfs領域の使用量は
/gpfs/usage_info/[アカウント名].txt
で確認することが出来ます。この情報は毎時0分に更新されます。
7: キュー構成:
2: 利用申請:
本システムの利用申請は、利用希望者が当該セメスターのHSC共同利用観測者
(PI+CoI、インテンシブを含む)かどうかで申請方法が異なります。
御自身が該当する方を御参照ください。
2-1: 利用申請(HSC共同利用観測者向け):
対象課題のPIには、セメスター開始前に、ハワイ観測所からLSCについての
ご案内をお送りします。
LSCの利用手順は、観測所からの案内をご参照ください。
また、HSC共同利用観測者向けのLSC利用に関するお問い合わせは、
ハワイ観測所のLSC担当(daikibo [at-mark] naoj.org)へお送りください。
2-2: 利用申請(一般向け):
随時利用申請を受け付けております。
利用申請ページ(一般向け)より申請をおこなってください。
3: 本システムの利用方法:
大規模解析システムは、計算資源をジョブ管理システムで管理します。
ユーザはログインノードにログインし、ジョブを投入することで
計算ノードで解析処理を実行可能です。
計算ノードへのログインや、システムに定められた目的以外の利用
およびジョブ管理システムを介さない利用は認めていません。
そのような利用を発見した場合には、予告なく強制ログアウト、
プロセスの強制停止を行いますのでご了承ください。
4: ログインノードへのログイン方法と利用上の注意:
天文台内からであれば、ログインノードへの ssh でログイン可能です。
ログインノードのFQDN: lsc.ana.nao.ac.jp
天文台外からであれば、NAOJ構成員用のVPNまたは多波長データ解析システムの
VPNで台内に接続後、 ログインノードへの ssh でログイン可能です。
アカウントは、多波長データ解析システムのアカウントを使用します。
(パスワードも同一)
大規模解析システムは、多波長データ解析システムのアカウント情報を共有しています。
ただし、これら登録情報の変更は大規模解析システム上で行うことが出来ません。
変更は、多波長データ解析システムで行うようにしてください。
2025年12月より、ログインノードの計算資源に以下の使用制限を導入しています。
- CPU使用率: ユーザ当たり200% (2コア フル稼働に相当)
- メモリ量: ユーザ全体で125G
ログインノードではジョブ投入やその準備など軽い処理のみ行なうこととし、多くの
計算資源を必要とする処理は計算ノードで行なうようにしてください。
5: ホーム領域:
ホーム領域には、200GBの使用制限(クォータ)がかかっています。
解析用のデータは、大容量作業領域(/gpfs)をご利用ください。
ホーム領域の使用量とクォータ情報は、ログインノードで
quota (-s)
の実行で確認可能です。(-sオプション: 量の単位を自動調整)
(領域名が/homeではなく「nas01:/mnt/nas/LUN_NAS」と表示される)
6: 大容量作業領域(/gpfs)の利用方法:
大容量作業領域(/gpfs)に、ご自身のアカウント名のディレクトリを
作成し、ご利用ください。
本領域は全ログイン・計算ノードから読み書き可能な領域で、30TB
の使用制限(クォータ)がかかっています(2025/6/2より)。
(定期)データ削除は「現状」では導入していませんが
今後の利用状況によっては導入の可能性があります。
現在のgpfs領域の使用量は
/gpfs/usage_info/[アカウント名].txt
で確認することが出来ます。この情報は毎時0分に更新されます。
7: キュー構成: